En este artículo realizaremos un ejercicio de clustering con ejemplo, utilizando el conjunto de datos del articulo: Spellman PT, et al. 1998. Comprehensive identification of cell cycle-regulated genes of the yeast Saccharomyces cerevisiae by microarray hybridization. Mol Biol Cell 9(12): 3273-97.
Nos vamos a descargar la matriz de expresión de 724 genes en diferentes condiciones y vamos a agruparlos por su similitud. Esto nos ayudará a descubrir patrones aplicando el algoritmo de aprendizaje no supervisado en machine learning. También aplicaremos un algoritmo de clustering para encontrar puntos de datos en común al utilizar el algoritmo.
Clustering con ejemplo
En este clustering con ejemplo tenemos el dataset de cómo se comportan las proteínas en el hongo de la levadura a lo largo del tiempo.
Lo que pretendemos ver es la correlación de las proteínas entre sí, es decir, de cómo cambian:
#Clustering con ejemplo
library (tidyverse)
spellman <- read.csv ("https://github.com/Bio723-class/example-datasets/raw/master/spellman-wide.csv", stringsAsFact [...])
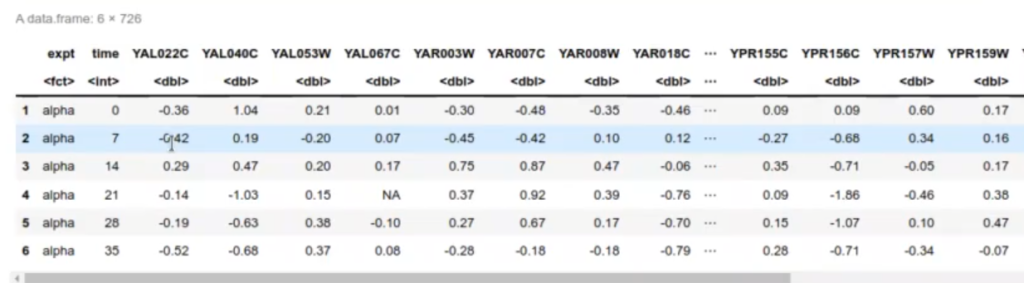
head (spellman)
#Clustering con ejemplo
summary (spellman)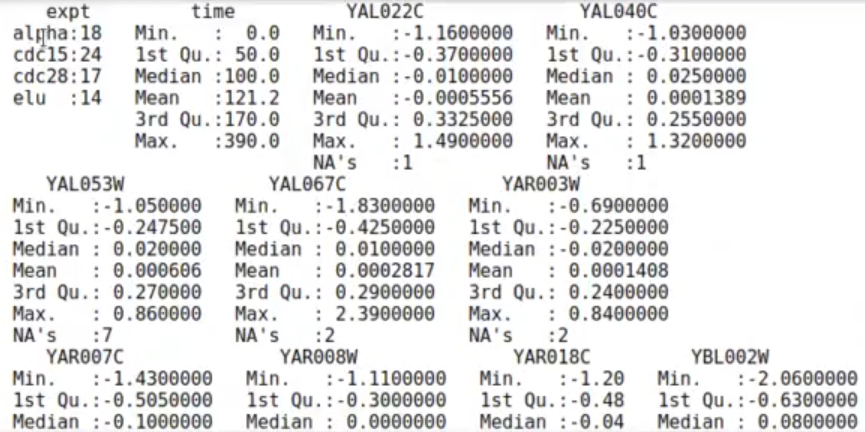
Ahora, realizaremos una matriz que indica la correlación de todos los pares con todos. Podríamos hacer esto de manera manual con el siguiente código:
#Clustering con ejemplo
abs (cor (spellman [ , c ( NC1, NC2)], use = "pairwise.complete.obs"))
#NC1/2: Número de columna (de proteína) 1 / 2 Pero en vez de eso, trazaremos una matriz que nos compara todos los resultados.
Cuanto más alta la correlación, mejor, porque más se parecen las muestras. Cuanto más baja la correlación, más separadas están las muestras. Por ello, debemos poner «1-» al inicio de la ecuación y quedaría «1- la correlación»; así, cuando la ecuación es cercana a 1, la distancia es prácticamente 0, y cuando la correlación es muy baja, la distancia es 1.
Esto significa que, en la matriz, los elementos que tienen un valor muy bajo (0.07, 0.2, etc.) están muy correlacionados entre sí.
#Clustering con ejemplo
1 - abs (cor (spellman [ , c ( -1, -2)], use = "pairwise.complete.obs"))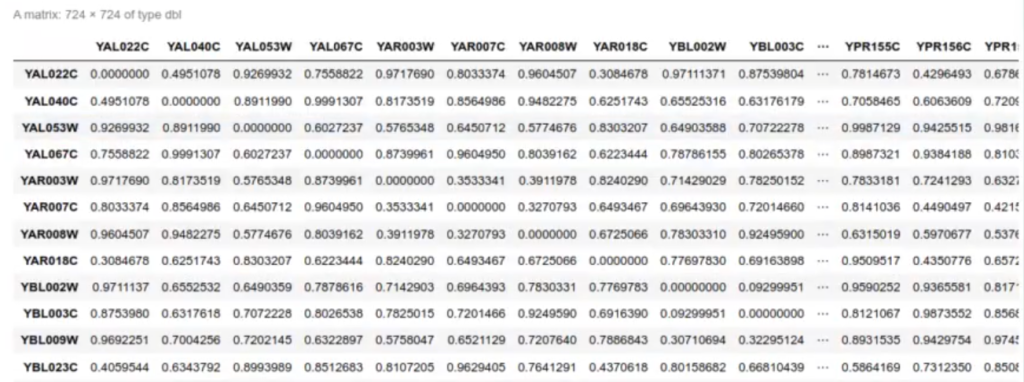
Aquí ya tenemos, entonces, la matriz de distancias, que ya no hay que calcular con el dist. El dist lo que hacía era calcular las distancias euclídeas, Manhattan o las que le dijéramos en todos los pares.
Las dos primeras columnas indican el tipo de experimento que se ha realizado. Las siguientes columnas indican el nivel de expresión genética del gen en cuestión, indicado por la columna. Por tanto, cada columna, a partir de la tercera, nos da los niveles de expresión de un gen en diferentes condiciones. Queremos encontrar los genes que muestran un comportamiento similar.
Como distancia ahora vamos a utilizar la correlación. Más concretamente: 1-cor. De esta forma, si dos genes tienen correlación muy cercana a 1, tendrán una distancia muy cercana a 0. A menor correlación, mayor distancia.
En la matriz anterior estamos mostrando directamente la matriz de correlación y de distancias y aquí estamos exponiendo el valor de campos de esa matriz.
#Clustering con ejemplo
spellman.cor <- cor (spellman [ , c (-1, -2) 1, use = "pairwise.complete.obs")
dim (spellman.cor)724 . 724
spellman.dist <- as.dist (1- abs (spellman.cor))
str (spellman.dist)‘dist’ num [1:261726] 0.495 0.927 0.756 0.972 0.803 . . .
– attr (*, «Labels») = chr [1:724] «YAL022C» «YAL040C» «YAL053W» «YAL067C»
– attr (*, «Size») = int 724
– attr (*, «call») = language as.dist.default (m = 1- abs (spellman.cor))
– attr (*, «Diag») = logi FALSE
– attr (*, «Upper») = logi FALSE
Ahora dibujaremos el dendograma para observar los clústeres que hay.
#Clustering con ejemplo
spellman.tree <- hclust (spellman.dist, method = "complete")
spellman.dend <- as.dendrogram (spellman.tree)
library(dendextend)
clusters <- cutree (spellman.dend, k = 4)
plot (color_branches (spellman.dend, k = 4), leaflab = "none")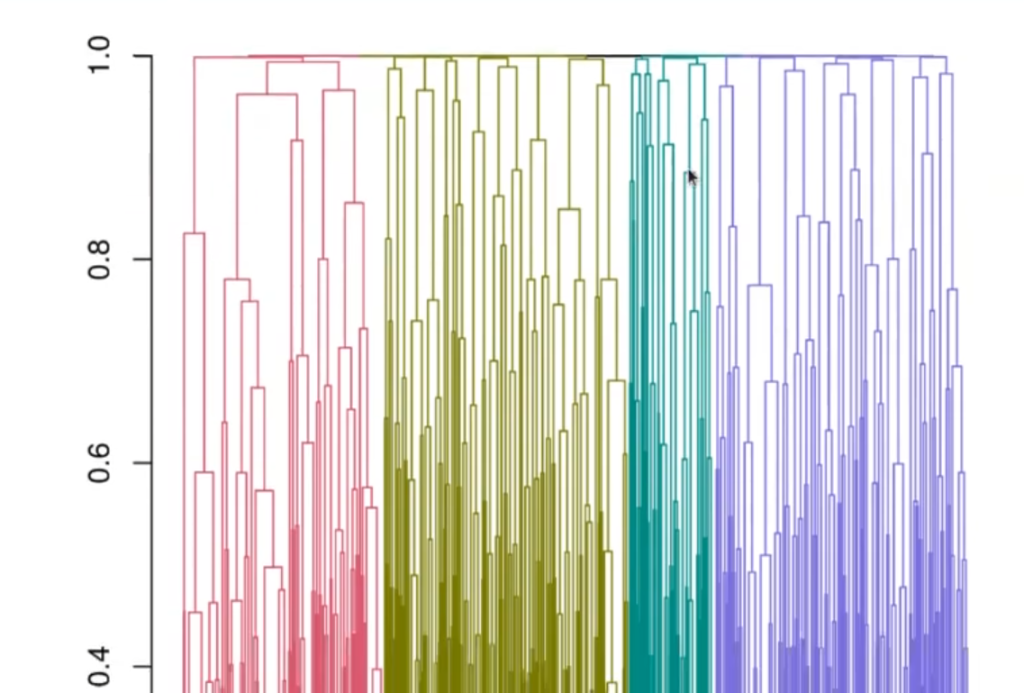
Cuando empiezan a aparecer las uniones desde muy arriba (como se puede observar en el dendrograma anterior), esto indica que a lo mejor hay mucho ruido y que va a ser difícil hacer grupos de correlación
A continuación, miramos las silhouettes:
library (cluster)
ss <- silhouette (clusters, spellman.dist)
plot (ss, col = 1 : max (clusters), border = NA)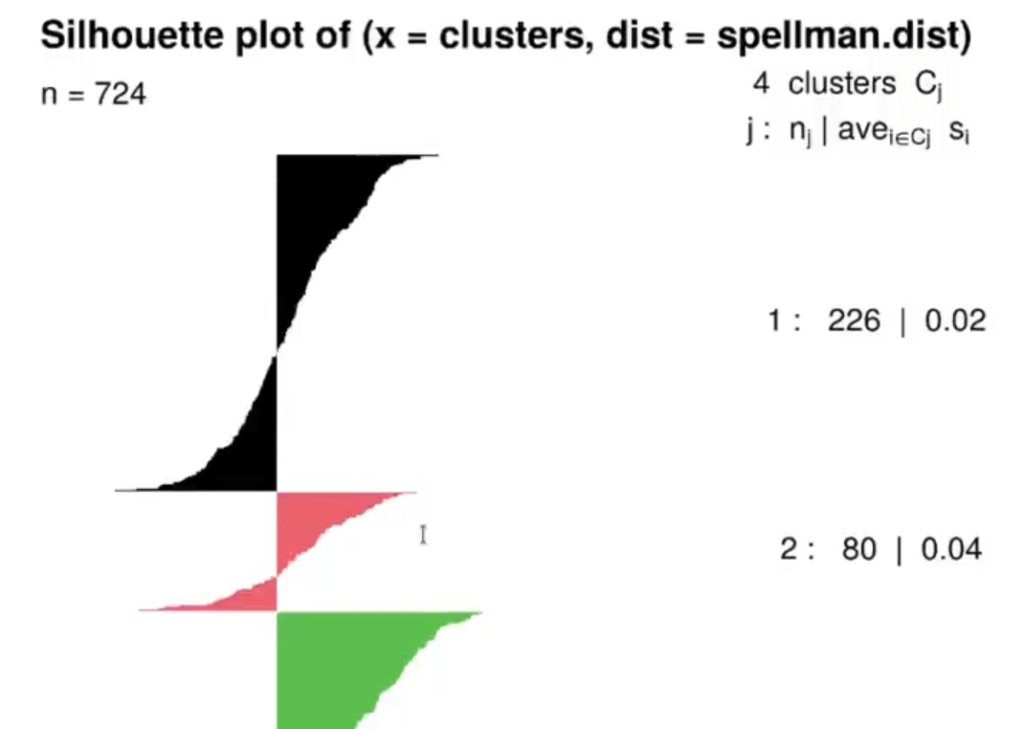
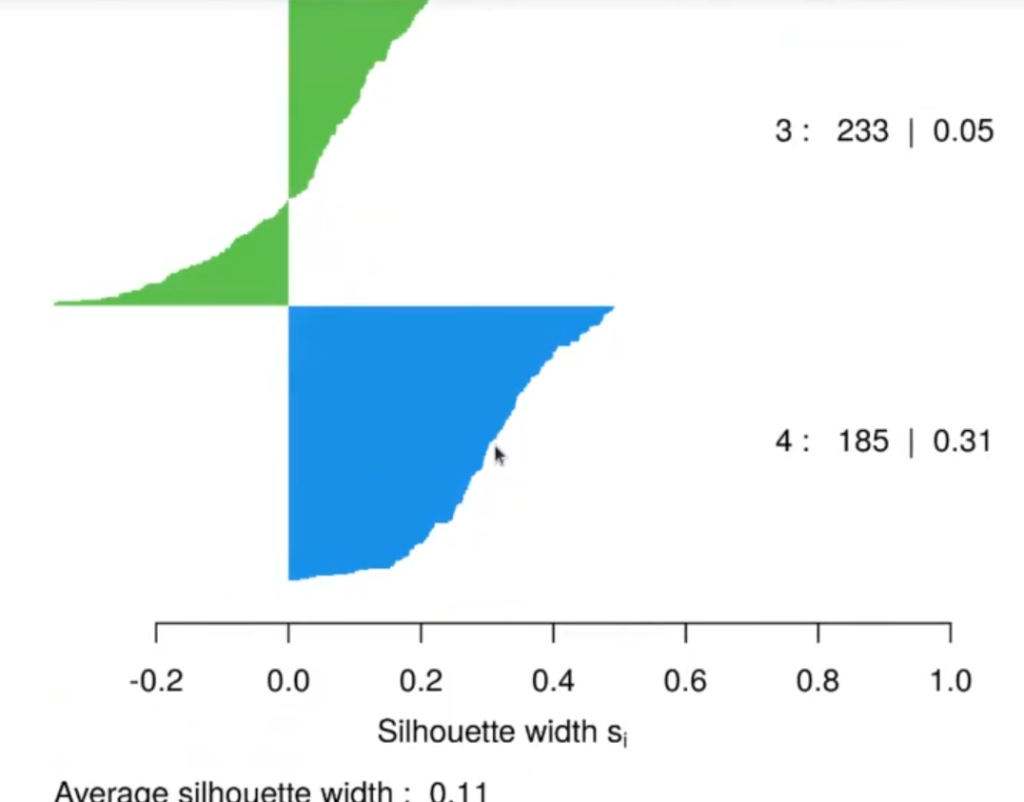
Vemos que las silhouettes tienen muchos valores negativos, aunque hay un clúster (el azul) que sí merece la pena. Habría que echarle un vistazo con más detenimiento a ese clúster azul que corresponde al número 4.
#Clustering con ejemplo
names (clusters [clusters == 4])
Por último, necesitaríamos un biólogo para que nos indicara qué relación hay entre estas proteínas y si tiene sentido que estén correlacionadas.
Ahora que hemos visto, una vez más, con un ejemplo, cómo funciona el clustering y sus grandes diferencias con el proceso de clasificación, podemos seguir aprendiendo. Para ello, Keepcoding tiene a tu disposición el Big Data y Machine Learning Full Stack Bootcamp, una formación intensiva en la que en pocos meses aprenderás todo lo necesario para incursionar en el mercado laboral IT. ¡Anímate a impulsar tu carrera y solicita más información!











