En este artículo veremos un ejemplo de clustering con el conjunto de datos de Iris (de Fisher o Anderson).
Este da las medidas en centímetros de las variables longitud y ancho del sépalo y longitud y ancho del pétalo, respectivamente, para 50 flores de cada una de las 3 especies de iris. Las especies son iris setosa, versicolor y virginica.
Ejemplo de clustering
Tenemos en este ejemplo de clustering distintas variables, la longitud de sépalo y el gancho. Vamos a ver si podemos hacer una clusterización con estos datos.
boxplot (iris)
summary (iris)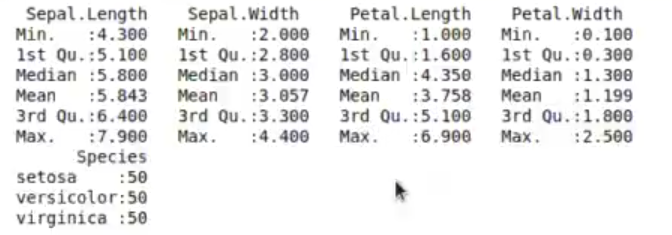
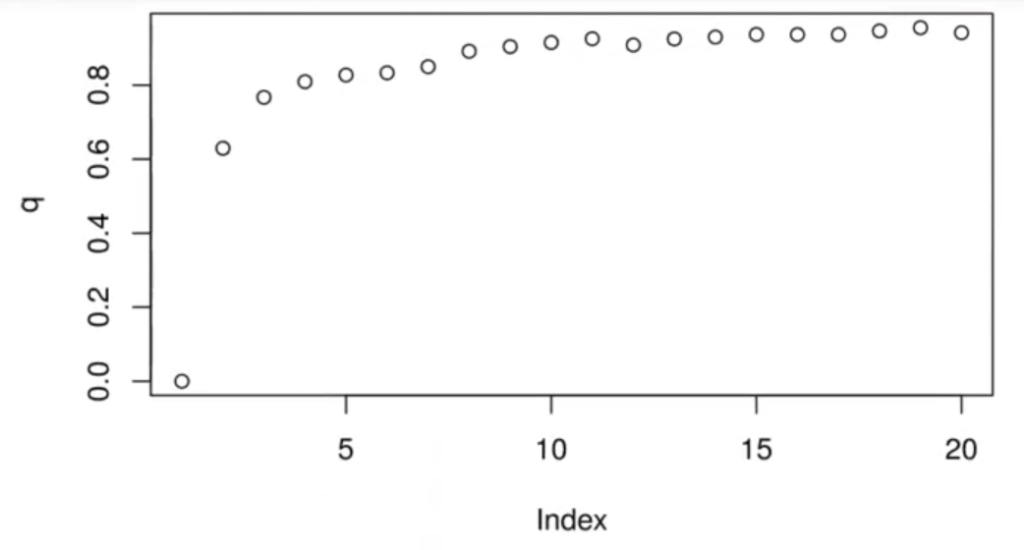
Lo que estamos pintando aquí es el betweenss entre el total, es decir el error, y lo máximo que podría ser es 1.
iris data <- iris [ , 1 : 4]
for (i in 1 : ncol (iris_data)) {
mn <- mean (iris_data [ , i], na.rm = T)
sd <- sd (iris_data [ , 1], na.rm = T)
iris_data [ , i ] <- (iris_data [ , i ] - mm) / sd
}
q <- c ()
for (k in 1 : 20) {
myclust <- kmeans (iris_data, k)
q [k] <- myclust$betweenss / myclust$totss
}
plot(q)Algo muy importante que hemos hecho con este ejemplo de clustering es estandarizar. Hemos restado la media y dividido por la desviación típica.
Aquí no estamos dividiendo entre train y testing, aunque también podría hacerse para calcular los betweenss. No obstante, en este caso no los utilizamos porque no es necesario y en realidad casi nunca se utiliza.
En esta segunda parte del ejemplo de clustering estamos mirando el número óptimo de clústeres. Hemos pintado clústeres de 1 hasta 20 y parece ser que el número óptimo está entre 2, 3 y 4 clústeres.
Pues bien, vamos a calcular tres clústeres y con ellos sale la solución;
myclust <- kmeans (iris_data, 3)
myclust$cluster3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 3 . 2 . 2 . 2 . 1. 1. 1. 2 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 2 . 1 . 1 . 1 . 1 . 2 . 1 . 1 . 1 . 1 . 2 . 2 . 2 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 2 . 2 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 1 . 2 . 1 . 2 . 2 . 2 . 2 . 1 . 2 . 2 . 2 . 2 . 2 . 2 . 1 . 1 . 2 . 2 . 2 . 2 . 1 . 2 . 1 . 2 . 1 . 2 . 2 . 1 . 2 . 2 . 2 . 2 . 2 . 2 . 1 . 1 . 2 . 2 . 2 . 1 . 2 . 2 . 2 . 1 . 2 . 2 . 2 . 1 . 2 . 2 . 1
Este es el resultado, el cual podemos pintar del siguiente modo:
cbind (iris, cl = myclust$cluster)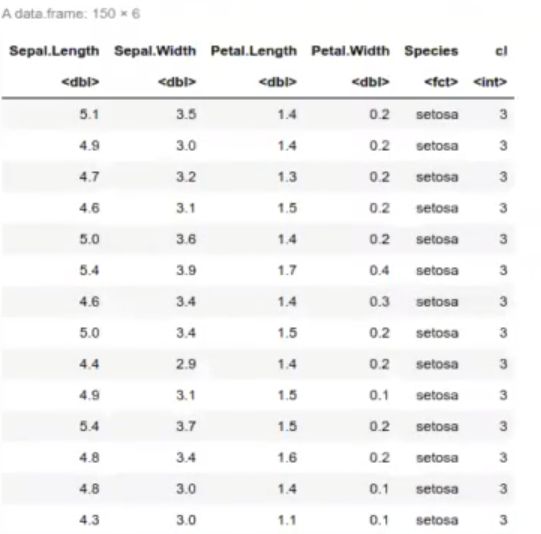
Lo que nos ha dicho esta tabla de ejemplo de clustering con el conjunto de datos Iris es que debemos tener cuidado porque esto, el iris, es solo para variables numéricas. Si queremos usarlo para variables categóricas, tenemos que definir una distancia para variables categóricas, entre otras opciones que existen para poder trabajar con variables categóricas con este dataset.
Los datos de la tabla nos los ha metido en el grupo 3, luego ha metido otros datos en el grupo 2 y así sucesivamente.
¿Qué significado tienen estos clústeres? Pues lo que podemos hacer en este ejemplo de clustering es darle significado con lo único que conocemos, que es la especie de iris.
table (iris$Species, myclust$cluster)| 1 | 2 | 3 | |
| setosa | 0 | 0 | 50 |
| versicolor | 39 | 11 | 0 |
| virginica | 14 | 36 | 0 |
Este ejemplo de clustering lo miramos con el nombre de la especie y vemos que para la setosa todos los datos nos los ha metido en el grupo 3; en cambio para la versicolor, la mayoría están en el grupo 1, y para la virginica, en el grupo 2.
Esto no es clasificación, no estamos diciéndole al algoritmo que nos clasifique. Le estamos diciendo que encuentre grupos de plantas que vea parecidas entre sí y que luego los compare con el nombre de la especie que conocemos.
Si quisiéramos hacer un clasificador, lo habríamos hecho entrenando.
Ahora que hemos visto un ejemplo de clustering ajustado al dataset Iris, podemos seguir aprendiendo sobre data mining. Para esto, KeepCoding tiene la mejor formación intensiva: Big Data, Inteligencia Artificial & Machine Learning Full Stack Bootcamp. Aquí, en pocos meses, adquirirás todos los conocimientos necesarios para incursionar en un mercado laboral lleno de oportunidades. ¡No esperes para solicitar más información!











