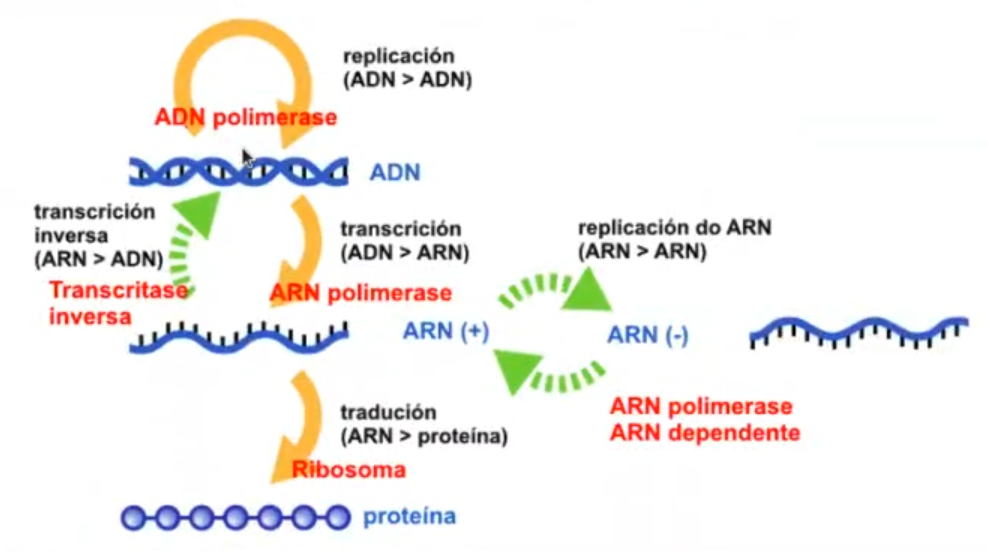
En este artículo realizaremos un ejercicio de preprocesado en R utilizando el dogma central de la biología molecular, una teoría que propone que la información genética fluye en una única dirección, del ADN al ARN y del ARN a la proteína, o del ARN a la proteína.
Ejercicio de preprocesado en R: proteínas
El conjunto de datos consiste en los niveles de expresión de 77 proteínas/modificaciones de proteínas que produjeron señales detectables en la fracción nuclear del córtex. Hay 38 ratones de control y 34 ratones trisómicos (síndrome de Down), para un total de 72 ratones. En los experimentos, se registraron 15 mediciones de cada proteína por muestra/ratón. Por lo tanto, para ratones de control hay 38×15 o 570 mediciones, y para ratones trisómicos hay 34 x15 o 510 mediciones. El conjunto de datos contiene un total de 1080 mediciones por proteína. Cada medida puede considerarse como una muestra/ratón independiente.
Las ocho clases de ratones se describen en función de características como el genotipo, el comportamiento y el tratamiento. Según el genotipo, los ratones pueden ser de control o trisómicos. Según el comportamiento, algunos ratones han sido estimulados para aprender y otros no; para evaluar el efecto del medicamento memantina en la recuperación de la capacidad de aprender en ratones trisómicos, algunos ratones han sido inyectado con la droga y otros no.
Clases:
- c-CS-s: ratones de control, estimulados para aprender, inyectados con solución salina (9 ratones) -CS-m: ratones de control, estimulados para aprender, inyectados con memantina (10 ratones).
- C-SC-s: ratones de control, no estimulados para aprender, inyectados con solución salina (9 ratones).
- c-SC-m: ratones de control, no estimulados para aprender, inyectados con memantina (10 ratones).
- t-CS-s: ratones con trisomia, estimulados para aprender, inyectados con solución salina (7 ratones).
- t-CS-m: ratones con trisomia, estimulados para aprender, inyectados con memantina (9 ratones).
- t-SC-s: ratones con trisomia, no estimulados para aprender, inyectados con solución salina (9 ratones).
- t-SC-m: ratones con trisomia, no estimulados para aprender, inyectados con memantina (9 ratones).
Los niveles absolutos de expresión de un gen no son comparables con los de otro. Para hacerlos comparables es necesario que todos los niveles de expresión se muevan en los mismos rangos.
Dicho todo esto, empecemos con nuestro ejercicio de preprocesado en R.
In [1] : mouse <- read.csv ("data/Data_Cortex_Nuclear.csv") #mouse_data <- mouse [ , c (2:78, 79)]
mouse data <- mouse [ , c (2:78, 82)]
head (mouse_data)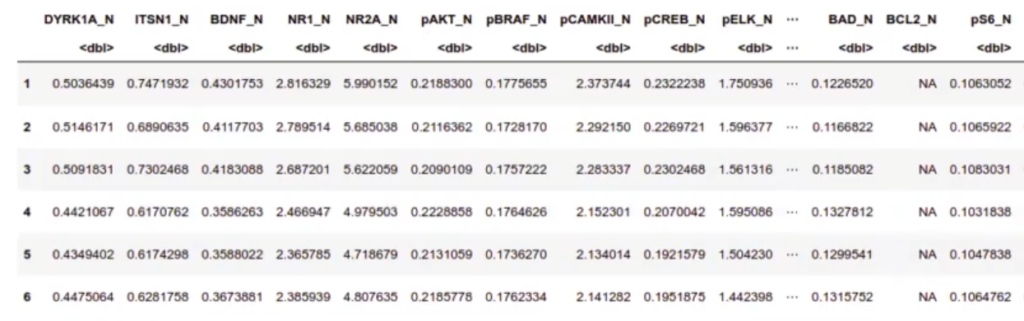
Continuando con el ejercicio de preprocesado en R, comprobamos que los márgenes en los que se mueve el nivel de expresión de cada gen es muy diferente y los hace difícilmente comparables.
In [21] : summary (mouse_data)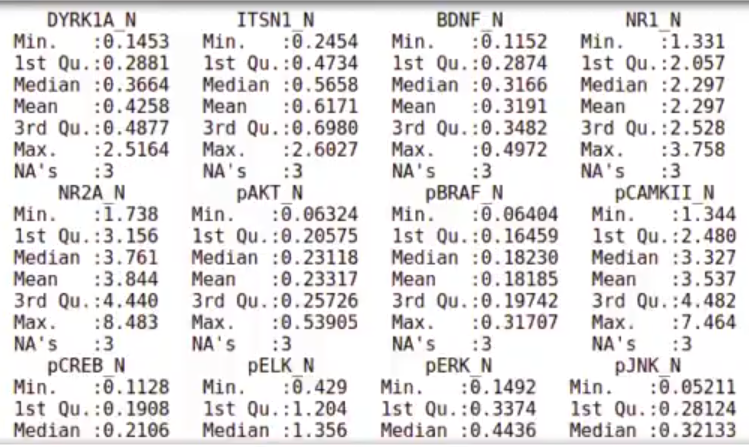
In [3] : library (ggplot2)
options (repr.plot.height = 4, repr. plot.width = 6, repr.plot.res = 200)
ggplot (mouse_data, aes (x = DYRKIA N, y = pCAMKII N, color = class)) + geom_point (size = 0.1) + coord_fixed()El resultado de esta parte del ejercicio de preprocesado en R es:
Warning message:
“Removed 3 rows containing missing values (geom_point).”
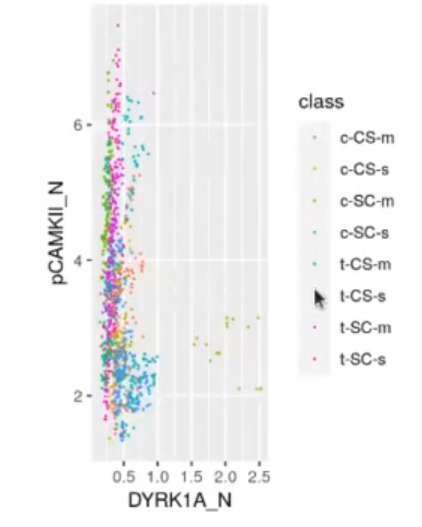
Tras aplicar una normalización en el ejercicio de preprocesado en R, podemos comparar su nivel de expresión con mayor claridad y ver qué influye en cada clase:
In [6] : library (pracma)
mouse data_noclass <- mouse data
mouse data noclass$class <- NULL
gem_m <- colMeans (mouse_data_noclass, na.rm = T)
gem_sd <- sapply (mouse_data_noclass, sd, na.rm=T)
mnCols <- repmat (gem_m, n = nrow (mouse_data_noclass), m = 1)
sdCols <- repmat (gem_sd, n = nrow (mouse_data_noclass), m = 1)
mouse_data_norm <- (mouse_data_noclass - mnCols) / sdCols mouse_data_norm$class <- mouse_data$classVamos a explicar un poco mejor el sapply. Este es equivalente a si cogemos los datos del ratón de la columna 1 (por ejemplo, puede ser la 2 o cualquier otra columna). Aplicamos el sd a la columna 1 y le pasamos el na.rm = T.
In [12] : sd (mouse_data_noclass[ , 1], na.rm=T)0.251640030326534
In [11] : head (sdCols[1, ])0.24936196351323
0.251640030326534
0.0493831216200576
0.347292908933713
0.933100065149151
0.0416344901350228
Los datos normalizados en el ejercicio de preprocesado en R al final son los datos originales (mouse_data_noclass) a los cuales se les resta la media (mnCols) y se dividen entre la desviación:
mouse_data_norm <- (mouse_data_noclass – mnCols) / sdCols
In [ ] : # Otra forma de hacer lo mismo utilizando funciones de R
mouse_data_noclass <- mouse_data
mouse_data_noclass$class <- NULL
mouse data norm <- as.data.frame (apply (mouse_data_noclass, 2, scale, center = TRUE, scale = TRUE))
mouse_data_norm$class <- mouse_data$class
In [ ] : summary (mouse_data_norm)
In [ ]: ggplot (mouse_data_norm, aes (x = DYRKIA_N, y = pCAMKII_N, color = class)) + geom_point (size = 0.1) + coord_fixed()Utilizaremos la función scale para hacer exactamente la misma normalización en el ejercicio de preprocesado en R:
?scale
Esta función nos sirve para centrar y escalar. Es decir, nos resta la media, nos centra y el escalar nos divide por la desviación típica.
Ahora que sabes cómo realizar un ejercicio de preprocesado en R de este estilo, puedes seguir realizando ejercicios que te ayudarán a afianzar tus conocimientos.
Si quieres algo más especializado, te recomendamos realizar nuestro Big Data, Inteligencia Artificial & Machine Learning Full Stack Bootcamp, en el cual, en muy poco tiempo, podrás aprender todo lo necesario para incursionar en el mercado laboral y obtener mejores oportunidades de trabajo. ¡No esperes para solicitar más información!







