Los valores no disponibles en R son valores para los cuales no tenemos ninguna medida en un conjunto de datos de una base de datos. Se representan con un NA.
Pertenecen a valores perdidos que no se han podido recuperar, errores de medida, perdidas de datos, etc.
Detección de valores no disponibles en R
Los valores no disponibles en R los detectamos con is.na(x). Podemos reemplazar su valor con la media, mediana, etc. o, simplemente, eliminarlos con na.omit(x).
Si estamos trabajando con series temporales, es posible que queramos hacer una interpolación de los valores perdidos:
- zoo::na.approx(x)
In [70] : data <- c (1, 3, NA, 6)
data1 . 3 . <NA> . 6
is.na (data)FALSE . FALSE . TRUE . FALSE
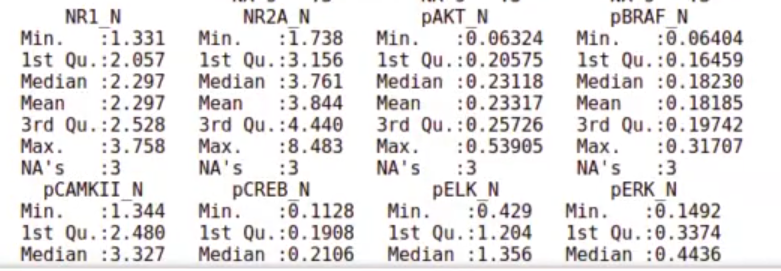
In [72] : mouse <- read.csv ("data/Data_Cortex Nuclear.csv")
#mouse data <- mouse [ , c (2:78, 79)]
summary (mouse)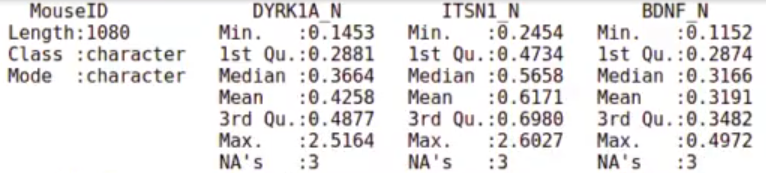
Una forma de rellenar estos datos es con, por ejemplo, la media.
Vamos a mirar el número de NA que hay por cada columna:
In [73] : sapply (mouse, function (x) sum (is.na (x)))
Con el which nos devuelve los valores que están en true:
In [74] : which (is.na (mouse$pMTOR_N))988 . 989 . 990
In [76] : nrow (mouse)1080
Inicialmente tenemos 1080 filas.
Para eliminar los NA utilizamos el na.omit. Pero cuidado, porque puede que haya muchos NA que no queramos eliminar, ya que si lo hacemos prácticamente nos quedaríamos sin datos. Debemos usar el na.omit con precaución para eliminar los valores no disponibles en R:
In [75] : mouse_no_na <- na.omit (mouse)
nrow (mouse_no_na)552
Ahora, después de eliminar los valores no disponibles en R, nos hemos quedado con 552.
In [77] : #Los índices que NO ha eliminado son:
length (na.action (na.omit (mouse)))528
In [78] : str (na.action (na.omit (mouse)))‘omit’ Named int [1:528] 1 2 3 4 5 6 7 8 9 10 …
– attr (* , «names») = chr [1:528] «1» «2» 3″ «4»…
In [79] : nrow (mouse) #número de filas totales
nrow (na.omit (mouse)) #número de filas que hay tras eliminar
nrow (mouse) - nrow (na.omit (mouse)) #número de filas eliminadas1080
552
528
También podemos observar las filas que hemos eliminado:
na.action (na.omit (mouse))
In [ ] : head (na.omit (mouse))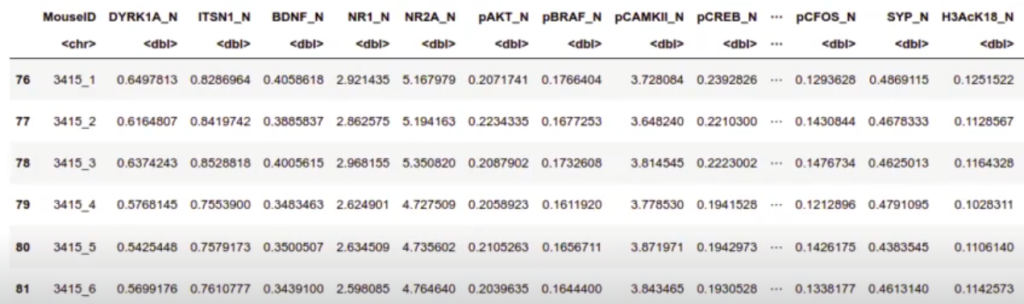
¿Qué hacer con los NA que no se eliminan?
Algunos de estos valores no disponibles en R simplemente se esfuman en ciertos modelos y no tienen ningún tipo de fallo. En cambio, existen modelos en los que sí se presentan fallos debido a los valores no disponibles en R que quedan en el dataset.
Algo que NO debemos hacer nunca es dejar los NA en 0. En vez de eso, podemos reemplazar dichos valores por la media de esa columna.
Vamos a ver tres formas distintas de hacer lo mismo:
In [83] : mouse_data <- mouse [ , 2:77]
In [84] : for (i in 1: ncol (mouse_data)){
mouse_data [ , i] <- replace (mouse_data [ , i] , is.na (mouse_data [ ,i]) , mean (mouse_data [ , i] , na.rm = T))
}Aquí podríamos reemplazar el elemento que es NA por la media. En caso contrario, dejamos el valor como está.
In [85] : mouse_data = as.data.frame (sapply (mouse_data, function (mcol) replace (mcol, is.na(mcol), mean (mcol, na.rm = TRUE))))Otra forma es con sapply. En lugar de usar un bucle, como en el ejemplo anterior, usamos el sapply y luego lo transformamos en un data frame. Este modo es ligeramente más rápido.
In [86] : library(zoo)
mouse_data = na.aggregate (mouse_data, FUN = mean)La última forma de reemplazar valores no disponibles en R (y la más fácil) es con na.aggregate. Esto lo que hace es agregar de la librería zoo todos los que son NA y los reemplazamos por las medias de cada columna.

Existen muchas otras funciones que se pueden usar para tratar con valores no disponibles en R, como el na.approx. Puedes descubrir qué hace el na.approx y muchísimo más en nuestro Big Data, Inteligencia Artificial & Machine Learning Full Stack Bootcamp, con el cual en unos pocos meses podrás aprender todo lo necesario para empezar en el mundo del Big Data para obtener mejores oportunidades laborales. ¡No esperes para solicitar más información y seguir el camino del éxito!











