En este articulo vamos a aplicar PCA a dataset Iris, un conjunto de datos con el que ya hemos trabajado en varias ocasiones y que es famoso porque muchos analistas lo usan en Big Data y sus derivados.
Aplicar PCA a dataset Iris
A continuación, te mostramos este proceso a través de un ejercicio práctico:
#aplicar PCA a dataset iris iris_data <- iris [ , 1 : 4] for (i in 1 : ncol (iris_data)) { mn <- mean (iris_data [ , 1 ], na.rm = T) sd <- sd (iris_data [ , i ], na.rm = T) iris_data [ , 1] <- (iris_data [ , i] - mn) / sd } #Utilizamos prcomp para calcular el pca priris <- prcomp (iris_data, center = FALSE, scale = FALSE) #Comprobamos que los dos primeros autovalores contienen aproximadamente el 90% de la varianza plot (priris$sdev ^ 2 / sum (priris$sdev ^ 2), main = "Autovalores")
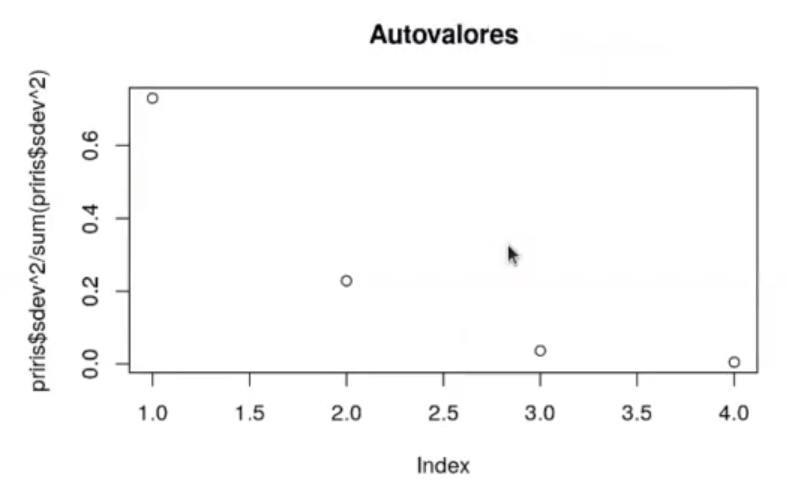
Observamos que apenas con dos elementos ya casi tenemos el 90% de la varianza: el primer elemento posee un 60 – 7 % (0.6+) y el segundo un 20% (0.2+).
Aquí lo que podemos hacer es aplicar PCA a dataset Iris.
Hacemos un k-means con tres clústeres:
#aplicar PCA a dataset iris number_of_clusters <- 3 number_of_pca <- 2 my_clust <- kmeans (priris$x [ , c (1 : number_of_pca)], number_of_clusters) plot (priris$x [ , c (1, 2)], col = my_clust$cluster) text (x = my_clust$centers [ , 1], y = my_clust$centers [ , 2], cex =1, pos = 4, labels = (1 : nrow (my_clust$centers)), col = "blue") table (iris$Species, my_clust$cluster)
| 1 | 2 | 3 | |
| setosa | 0 | 0 | 50 |
| versicolor | 11 | 39 | 0 |
| virginica | 36 | 14 | 0 |
El resultado es lo que se muestra a continuación:
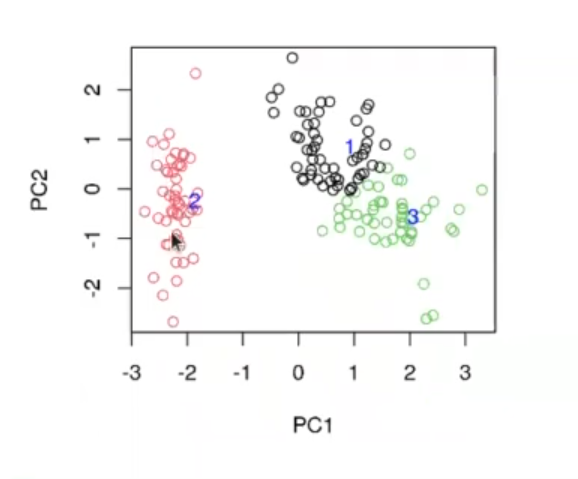
Hemos cogido las 4 dimensiones que teníamos de sépalos y pétalos (el alto y el ancho de ambas) y lo hemos dividido en 2. Nos han salido dos grupos. El grupo 1 corresponde a los datos pintados en color rojo y al grupo 2 pertenecen los datos color negro y verde.
El algoritmo ha hecho un intento por diferenciar este último grupo y dividirlo en dos, pero ha sido un intento fallido.
En lugar de hacer un clúster, a lo mejor podríamos pintar las distintas especies por color. Si usamos la librería dbscan, solo encontrará dos grupos:
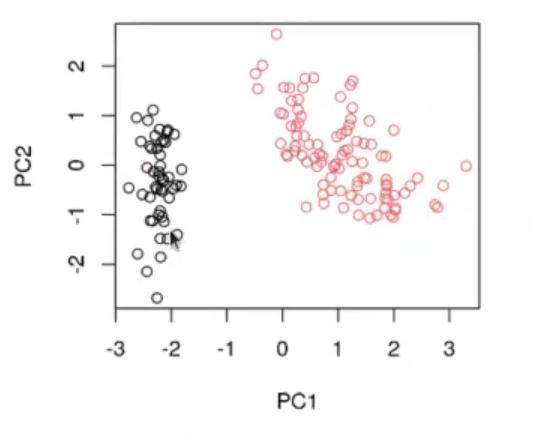
#aplicar PCA a dataset iris library (dbscan) number_of_pca <- 2 #my_clust <- hdbscan (priris$x [ , c (1 : number_of_pca)], minPts = 4) my_clust <- dbscan (priris$x [ , c (1 : number_of_pca)], minPts = 5, eps = 0.8) plot (priris$x[ , c (1, 2)], col = my clust$cluster) table (iris$Species, my_clust$cluster)
| 0 | 1 | 2 | |
| setosa | 1 | 49 | 0 |
| versicolor | 0 | 0 | 50 |
| virginica | 3 | 0 | 47 |
Los dos grupos que nos encuentra se muestran a continuación:
Ahora realizaremos la matriz de rotación.
Recordemos que estas matrices solo nos dicen qué tenemos que hacer cuando nos venga una muestra, cómo transformarla al dominio anterior.
Cuando, por ejemplo, nos llegue una nueva flor con una longitud de sépalo, lo multiplicamos por el PC1. Esto lo hacemos con el largo y ancho del sépalo y del pétalo. Lo que nos queda es un valor en la coordenada del PC1.
Lo mismo haríamos con el PC2, con el PC3 y así sucesivamente.
#aplicar PCA a dataset iris
priris$rotation| PC1 | PC2 | PC3 | PC4 | |
| Sepal.Length | 0.5210659 | -0.37741762 | 0.7195664 | 0.2612863 |
| Sepal.Width | -0.2693474 | -0.92329566 | -0.2443818 | -0.1235096 |
| Petal.Length | 0.5804131 | -0.02449161 | -0.1421264 | -0.8014492 |
| Petal.Width | 0.5648565 | -0.06694199 | -0.6342727 | 0.5235971 |
#aplicar PCA a dataset iris
head (priris$x)
head (iris)| PC1 | PC2 | PC3 | PC4 |
| -2.257141 | -0.4784238 | 0.12717962 | 0.024087508 |
| -2.074013 | 0.6718827 | 0.23382552 | 0.102662845 |
| -2.356335 | 0.3407664 | -0.04405390 | 0.028282305 |
| -2.291707 | 0.5953999 | -0.09098530 | -0.065735340 |
| -2.381863 | -0.6446757 | -0.01568565 | -0.035802870 |
| -2.068701 | -1.4842053 | -0.02687825 | 0.006586116 |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species | |
| <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | |
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 6 | 5.4 | 3.9 | 1.7 | 0.4 | setosa |
¿Te ha gustado este tuto?
Hemos visto cómo aplicar PCA a dataset Iris, un conjunto de datos que es bastante familiar para todos aquellos que hayan estudiado algo relacionado con el Big data, en especial la analítica.
Si quieres seguir formándote, puedes hacerlo de forma íntegra e intensiva por medio de nuestro Big Data, Inteligencia Artificial & Machine Learning Full Stack Bootcamp, con el que adquirirás todos los conocimientos relacionados con el área en pocos meses, a nivel teórico y práctico y con la guía constante de grandes profesionales en el área.
¿Quieres darle un impulso a tu carrera profesional y triunfar en el mercado laboral IT? ¡Anímate a cambiar tu vida y solicita ahora más información!











