En este artículo realizaremos un ejemplo de regresión logística con PCA por medio de un dataset que muestra tejidos de diferentes tumores de mama. Algunos son benignos y otros malignos.
Regresión logística con PCA: ejemplo
Vamos a hacer un clasificador para diferenciarlos:
#regresión logística con PCA
library (pracma)
library (dplyr)
set.seed (1234)
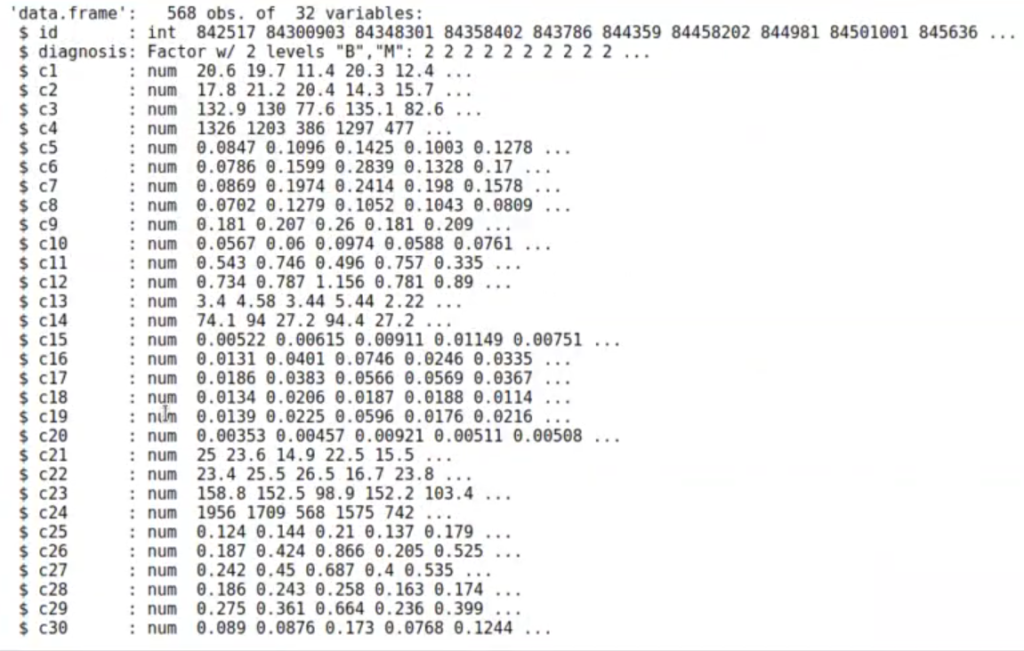
wdbc <- read.csv ("data/wdbc.data", col.names = c ("id", "diagnosis", paste0 ("c", 1 : 30)), stringsAsFactor = T)
str (wdbc)
wdbc <- wdbc %>% select (-id)
idx <- sample (1 : nrow (wdbc), round (nrow (wdbc) * 0.7))
wdbc.train <- wdbc [ idx, ]
wdbc.test <- wdbc [ -idx, ]
prwdbc <- prcomp (wdbc.train %>% select (-diagnosis), center = TRUE, scale. = TRUE)
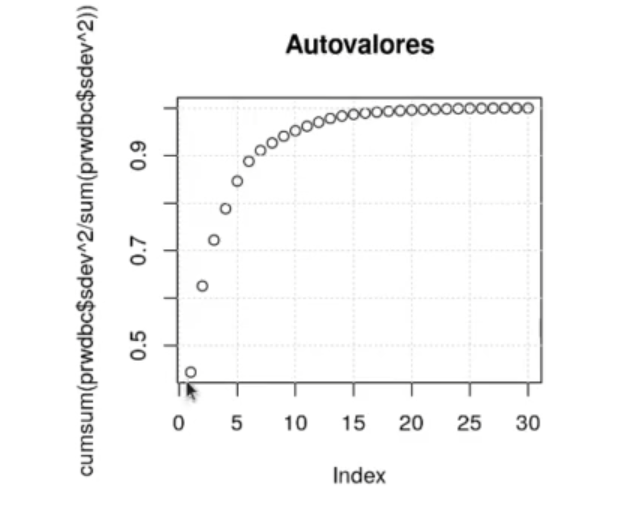
#Comprobamos que los 5 primeros autovalores contienen aproximadamente el 90% de la varianza
plot (cumsum (prwdbc$sdev ^ 2 / sum (prwdbc$sdev ^ 2)), main = "Autovalores")
grid ()
wdbc.train_pca <- as.data.frame (prwdbc$x [ , 1 : 2])
wdbc <- wdbc %>% select ( -id ) idx <- sample (1 : nrow (wdbc), round (nrow (wdbc) * 0.7)) wdbc.train <- wdbc [ idx, ] wdbc.test <- wdbc [ -idx, ] dim (wdbc.train)
398 31
#regresión logística con PCA prwdbc <- prcomp (wdbc.train %>% select (-diagnosis), center = TRUE, scale. = TRUE) #Comprobamos que los 5 primeros autovalores contienen aproximadamente el 90% de la varianza plot (cumsum (prwdbc$sdev ^ 2 / sum (prwdbc$sdev ^ 2)), main = "Autovalores") grid ()
wdbc.train_pca <- as.data.frame (prwdbc$x [ , 1 : 2])
wdbc.train_pca$diagnosis <- wdbc.train$diagnosis
summary (wdbc.train_pca)
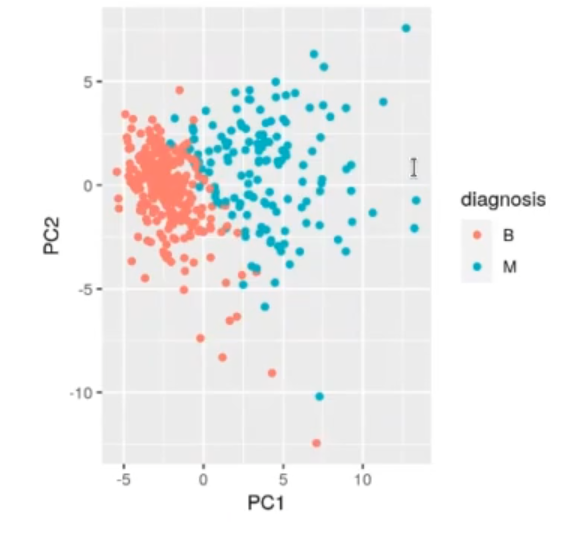
ggplot (wdbc.train_pca, aes (x = PC1, y = PC2, color = diagnosis)) + geom_point ()
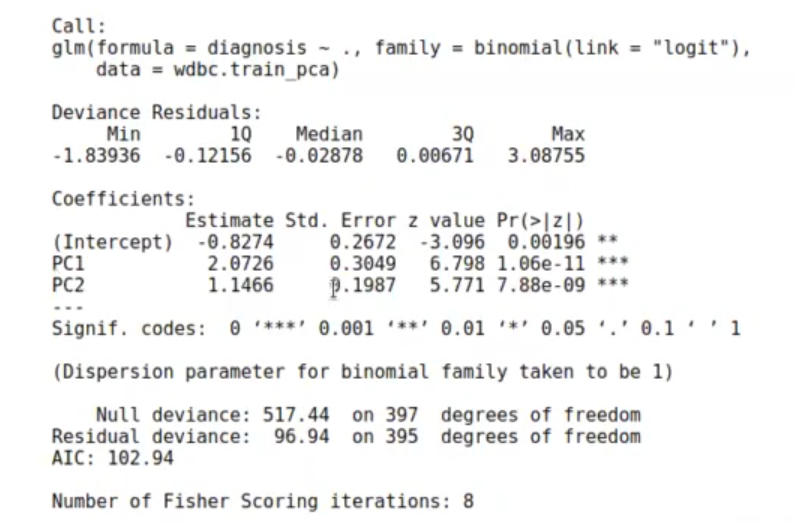
model_wdbc <- glm (data = wdbc.train_pca, formula = diagnosis~., family = binomial (link = 'logit'))
summary (model_wdbc)
y_predict <- predict (model_wdbc, wdbc.train_pca)
y_factor <- as.factor (ifelse (y_predict < 0, "B", "M"))
table (real = wdbc.train$diagnosis, pred = y_factor)

predict (prwdbc, wdbc.test %>% select (-diagnosis)) [ , c ("PC1", "PC2")]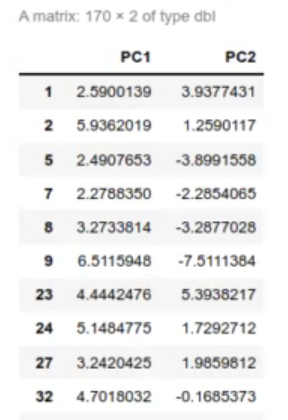
#regresión logística con PCA
wdbc.test_pca <- as.data.frame (predict (prwdbc, wdbc.test %>% select (-diagnosis))[ , c ("PC1", "PC2")])
summary (wdbc.test_pca)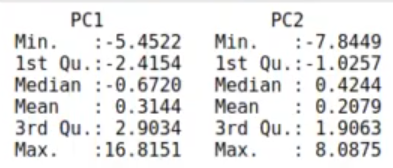
y_predict <- predict (model_wdbc, wdbc.test_pca)
y_factor <- as.factor (ifelse (y_predict < 0, "B", "M"))
table (real = wdbc.test$diagnosis, pred = y_factor)
cf_m <- caret :: confusionMatrix (data = y_factor, reference = wdbc.test$diagnosis, positive = "M")
cf_m
paste ("La precisión es:", cf_m$table [2, 2] / sum (cf_m$table [2, ]))
paste ("La exhaustividad (recall, sensitivity) es:", cf_mstable [2, 2] / sum (cf_mstable[ , 2]))
paste ("La exactitud (accuracy) es:", (cf_mstable [2, 2] + cf_mstable [1, 1]) / sum (cf_mstable))
bnt_test = binom.test (cf_mstable [2, 2] + cf_mstable [1, 1], sum (cf_mstable))
paste ("El intervalo de confianza de la exactitud es: [".paste0 (bnt_testsconf.int, collapse = ","), "]")
library (ROCR)
p <- predict (model_wdbcwdbc.test_pca, type = "response"}
pr <- prediction (p, wdbc.test$diagnosis)
prf_auc = performance (pr, measure = "auc")
paste ("The AUC is", [email protected][[1]])
prf <- performance (pr, measure "tpr", x.measure = "fpr")
plot (prf)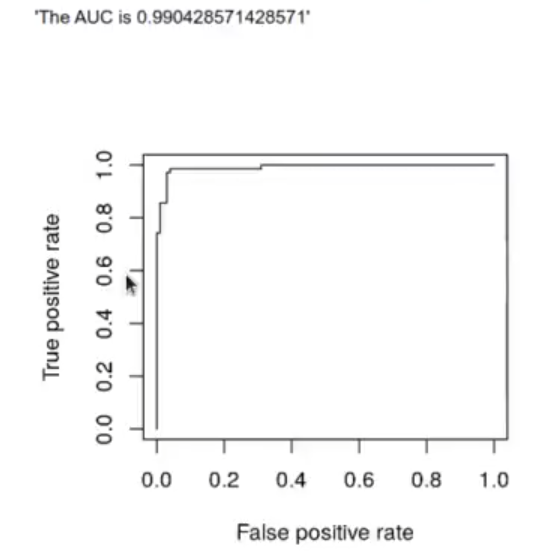
wdbc.train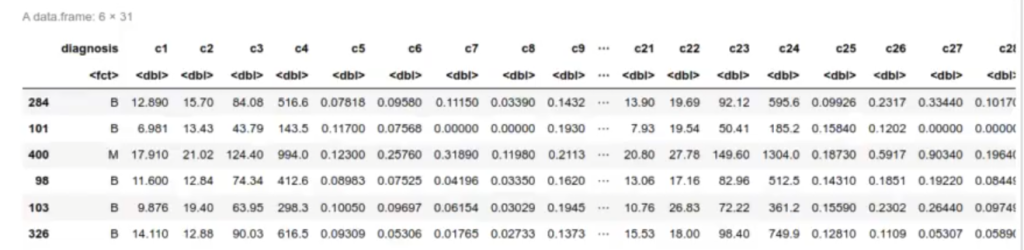
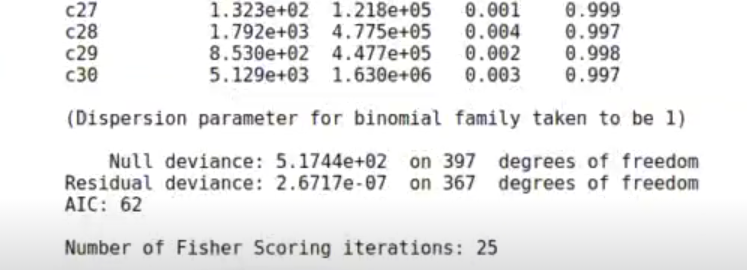
model _wdbc_lin <- glm (data = wdbc.train, formula = diagnosis~., family = binomial (link = 'logit'))
summary (model_wdbc_lin)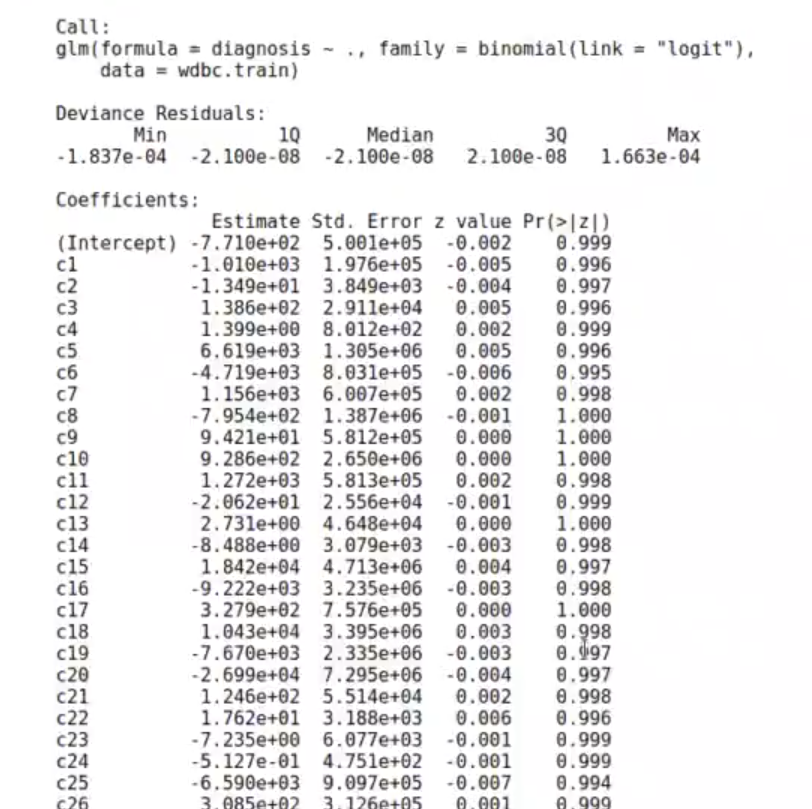
y_predict <- predict (model_wdbc_lin, wdbc.train)
y_factor <- as.factor (ifelse (y_predict < 0, "B", "M"))
table (real = wdic.trainsdiagnosis, pred = y_factor)
y_predict <- predict (model_wdbc_lin, wdbc.test)
y_factor <- as.factor (ifelse (y_predict < 0, "B", "M"))
table (real = wdbc.test$diagnosis, pred = y_factor)Si te ha gustado la temática de este pos, recuerda que puedes seguir aprendiendo con nuestro Full Stack Bootcamp en Big Data, una formación intensiva en donde aprenderás todo lo necesario para incursionar en el mundo laboral. ¡Solicita más información y empieza a cambiar tu vida!











