Entre las muchas cosas que se puede hacer en este inmenso panorama, podemos hacer una predicción del covid con Big Data. Fue algo tremendamente útil en sus primeras etapas, donde se buscaba evaluar el impacto mundial que este virus tendría para, de este modo, tomar decisiones gracias a la inteligencia artificial y realizar modelos de predicción con los datos disponibles.
¿Qué encontrarás en este post?
Toggle¿Cómo hacer una predicción del covid con Big Data?
Vamos a ver los datos que teníamos previamente y la serie acumulada para poder hacer la predicción del covid con Big Data:
library(tidyverse)
#df_acumulados <- read.csv ("https://covid19.isciii.es/resources/ serie_historica_acumulados.csv")
df_acumulados <- read.csv ("data/serie historica_acumulados.csv")
df_acumulados$FECHA <- as.Date (df_acumulados$FECHA, format = "% d / % m / % Y")
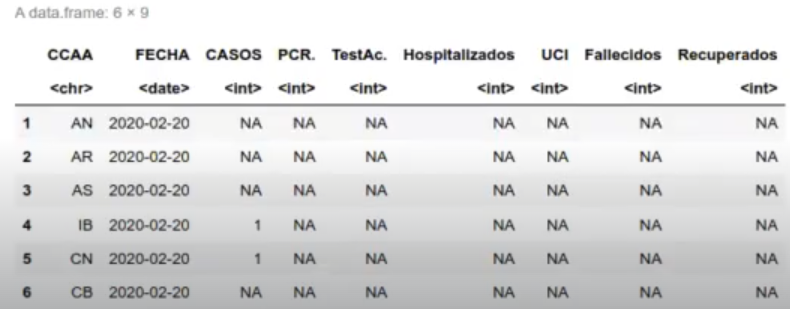
head (df_acumulados)
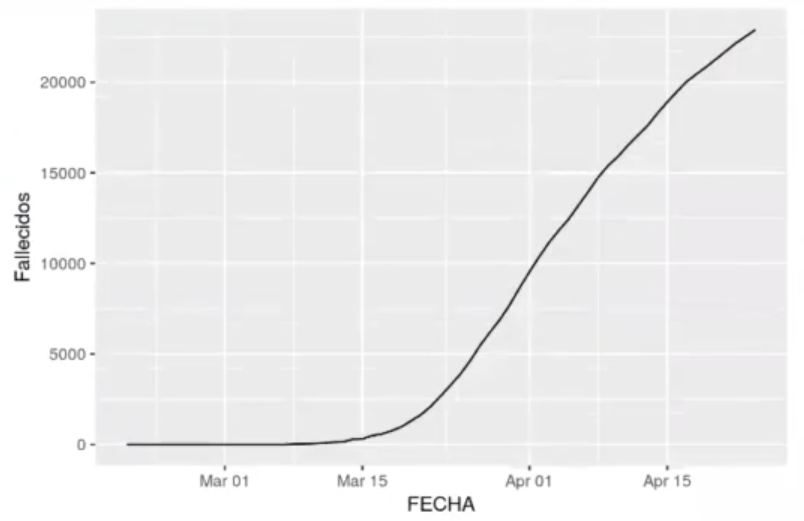
Vamos a hacer la predicción del covid con Big Data sobre el total de fallecidos. Así pues, mostraremos el acumulado de los fallecidos:
df_acumulados_all <- df_acumulados %>% group_by (FECHA) %>%
summarise_at (c ("CASOS", "Hospitalizados", "UCI", "Fallecidos", "Recuperados"), sum, na.rm= TRUE)
ggplot (df_acumulados all, aes (x = FECHA, y = Fallecidos)) + geom_line()Warning message:
«Removed 1 row(s) containing missing values (geom_path).»
Filtramos los datos con fecha a partir del 1 de abril de 2020 para observar la parte exponencial de la gráfica anterior, porque una vez que empezó el confinamiento, la curva se fue doblegando.
df_acumulados <- df_acumulados %>% filter (FECHA < as.Date ("2020 - 04 - 01"))
df_acumulados_all <- df_acumulados all %>% filter (FECHA < as.Date ("2020 - 04 - 01"))
df_acumulados_all$Fallecidos_dia <- c (NA, diff (df_acumulados_all$Fallecidos))
df_acumulados_all$dia <- seq (nrow (df_acumulados_all))
tail (df_acumulados_all)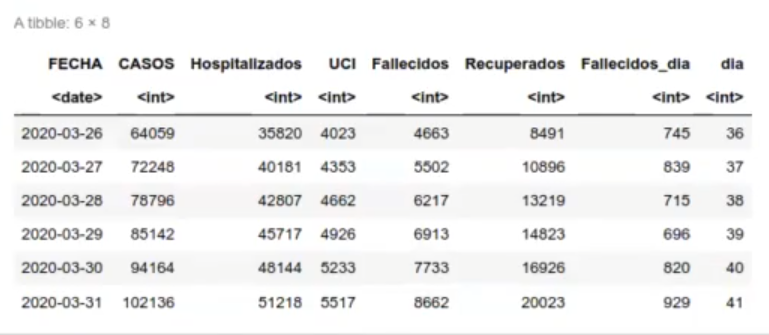
Para hacer la predicción del covid con Big Data, primero vamos a hacer el modelo de Poisson, en el cual se calcula el número de fallecidos en función del día.
Aquí ya no estamos calculando los acumulados, sino la diferencia de fallecidos:
model_poisson <- glm (df_acumulados_all, formula = Fallecidos_dia ~ dia, family = poisson)
summary (model_poisson)
Nos sale 0.667573 y 0.157938. ¿Qué significa esto? Pues que cada día las víctimas se multiplicaron un 17 %, que es el exponente de 0.157938. La línea de este último valor equivale al número de víctimas que se multiplicaron por día.
paste ("Cada día las víctimas se multiplican por", exp (model_poisson$coefficients [2]))‘Cada día las víctimas se multiplican por 1.17109336753171’
Pintamos este valor en una gráfica:
preds <-df_acumulados_all [ , c ("Fallecidos_dia", "dia", "FECHA")]
preds$pred <- predict (mode_poisson, preds, type = "response")
#preds$pred <- exp (model_poisson$_coefficients [2] preds$dia + model_poisson$coefficients [1])
ggplot (preds, aes (x = FECHA)) +
geom_line (aes (y = Fallecidos_dia, color = "Real")) +
geom_line (aes (y =pred, color = "Estimado"))Warning message:
«Removed 1 row(s) containing missing values (geom_path).»
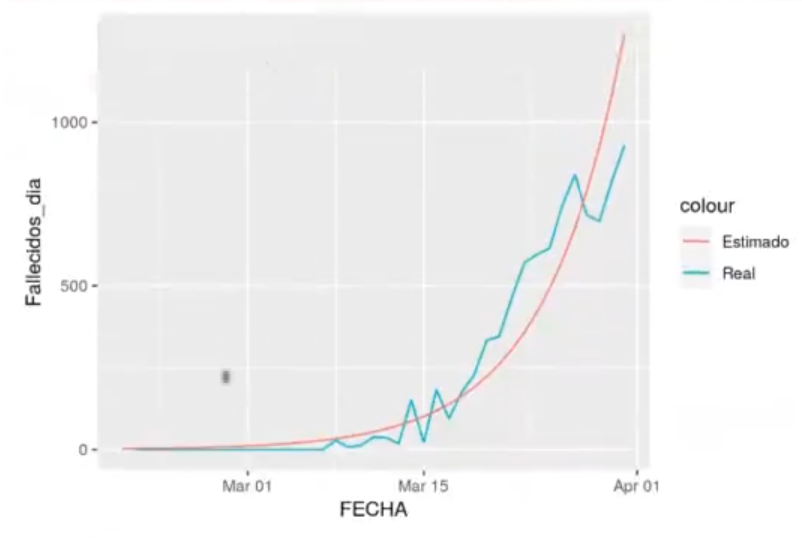
En esta predicción del covid con Big Data vemos cómo es el valor estimado y cómo es el valor real.
Regresión lineal sobre logaritmos
En lugar de aplicar el logaritmo en una función exponencial, podemos utilizar una regresión lineal sobre los logaritmos para hacer esta predicción del covid con Big Data:
df_acumulados_all$log_fallecidos <- log (df_acumulados_all$Fallecidos_dia)
df_acumulados_all_no_inf <-df_acumulados_all [ ! is.infinite (df_acumulados_all$log_fallecidos),]
model <- lm (data = df_acumulados_all_no_inf,
formula = log_fallecidos ~ dia)
summary (model)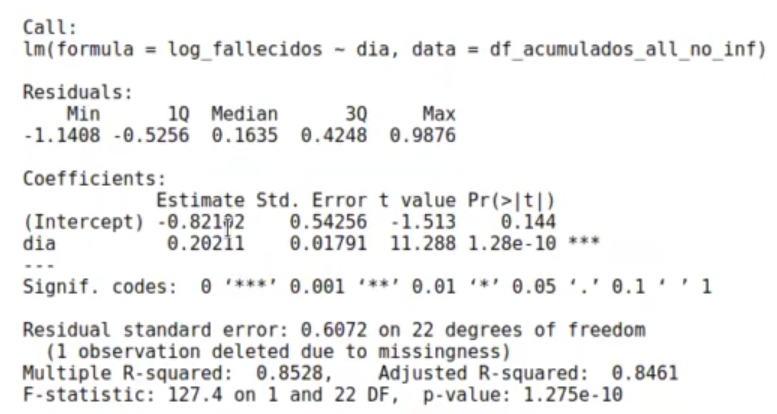
paste ("Cada día las víctimas se multiplican por", exp (model$coefficients [2]))‘Cada día las víctimas se multiplican por 1.22398743142683’
preds <- df_acumulados_all [ , c ("Fallecidos_dia", "log_fallecidos", "dia", "FECHA")]
preds$pred <- predict (model, preds)
preds$pred_fallecidos <- exp (preds$pred)
ggplot (preds, aes (x = FECHA)) +
geom_line (aes (y = Fallecidos_dia, color = "Real")) +
geom_line (aes (y = pred_fallecidos, color = "Estimado"))
Warning message:
«Removed 1 row(s) containing missing values (geom_path).»
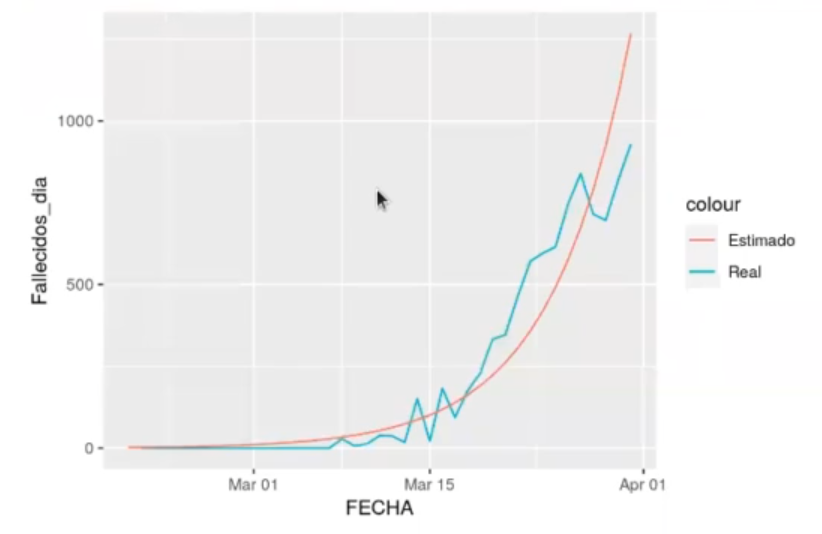
¿Qué sigue?
Recuerda que si te ha interesado este tema de la predicción del covid con Big Data y quieres seguir aprendiendo sobre el análisis de datos, puedes solicitar información para inscribirte en nuestro Bootcamp Full Stack Big Data, Inteligencia Artificial & Machine Learning. En muy pocos meses dominarás todos los conocimientos necesarios para incursionar en el mercado laboral más prolífico del momento. ¿A qué estás esperando? ¡Anímate y escríbenos!








